Til n’existe pas de modèle génétique unique pour le cancer. Au lieu de cela, chaque cancer individuel s’appuie sur un ensemble de mutations acquises qui confèrent aux cellules un avantage sélectif et des tactiques supérieures d’évasion immunitaire et de prolifération. Grâce aux technologies de séquençage de nouvelle génération, de nombreux patients diagnostiqués avec un cancer particulier peuvent découvrir si leurs tumeurs abritent des mutations spécifiques qui les rendent plus sensibles à des thérapies particulières. Cependant, les approches ciblées ne parviennent pas à capturer l’ensemble des altérations et des biomarqueurs nichés dans l’architecture génétique complexe de la tumeur d’un patient, obscurcissant potentiellement le meilleur plan de traitement disponible pour un patient individuel.
Dans une étude publiée dans Médecine naturelleles chercheurs ont développé un pipeline bioinformatique pour intégrer les données de séquençage du génome entier (WGS) de 13 880 tumeurs avec les données cliniques correspondantes des patients.1 L’étude à grande échelle a révélé des mutations somatiques et germinales de l’ADN qui affectent le pronostic, soulignant l’influence potentielle de la génomique complète du cancer sur les résultats pour les patients.

Mark Caulfield, aujourd’hui chercheur en médecine génomique à l’Université Queen Mary de Londres, était auparavant scientifique en chef de Genomics England, où il a supervisé le projet 100 000 Genomes. Il a reçu le titre de chevalier en 2019 pour son travail sur le projet.
Université Queen Mary de Londres
« Le cancer est une maladie causée par des génomes désordonnés », a déclaré Marc Caulfield, chercheur en médecine génomique à l’Université Queen Mary de Londres et co-auteur de l’étude. Bon nombre des accidents génétiques qui apparaissent ne sont pas pertinents pour la croissance du cancer, mais d’autres contiennent des informations qui permettraient aux chercheurs de prédire la progression et les vulnérabilités de la tumeur. Caulfield et son équipe se sont mis à la recherche de ces empreintes génétiques riches en informations.
Pour de nombreux cancers, les tests génomiques visant à identifier des variantes génétiques spécifiques, comme les polymorphismes mononucléotidiques, font déjà partie des soins standard. Cependant, ces panels ciblés ont une portée limitée. « (Une approche globale) vous donne une image complète de ce qui se passe dans la tumeur d’un patient individuel et que vous pourriez manquer si vous finissez par utiliser des panels ciblés », a déclaré Arul Chinnaiyan, chercheur en cancérologie à l’Université du Michigan qui n’a pas participé à l’étude. « Cette étude met vraiment en évidence l’importance de cela. »
Pour explorer les gènes qui façonnent la santé et la maladie, le gouvernement britannique a créé le Projet 100 000 génomes en 2013.2 Dirigés par la société publique Genomics England, des chercheurs ont entrepris de séquencer le génome de patients au sein du National Health Service (NHS), le système de santé financé par l’État du Royaume-Uni. Dans le cadre du programme de lutte contre le cancer de l’initiative, chercheurs et cliniciens se sont associés pour identifier les changements génétiques qui conduisent à la progression du cancer. Dans la présente étude, les chercheurs ont séquencé les tumeurs des patients ainsi que leur ADN germinal correspondant. Analyser l’ADN normal et hérité d’un patient aux côtés de l’ADN tumoral peut fournir une image plus grande de susceptibilité à d’autres cancers ou informer les cliniciens sur les traitements les plus appropriés pour le patient compte tenu des variantes génétiques affectant le métabolisme des médicaments.3
Les 13 880 échantillons couvraient 33 types de tumeurs. En un seul test, WGS peut détecter de petites variantes somatiques, notamment des variantes mononucléotidiques (SNV), des insertions et des délétions, ainsi que des aberrations du nombre de copies (CNA). En intégrant des données cliniques longitudinales réelles, les chercheurs pourraient évaluer le rôle de ces facteurs génomiques dans la réponse au traitement et la survie.
« L’étendue de l’étude était assez impressionnante », a déclaré Chinnaiyan.
Caulfield et son équipe ont commencé par examiner les taux de mutation des gènes inclus dans la version la plus récente du National Genomic Test Directory for Cancer (NGTDC), une liste qui précise à quels panels génomiques un patient est éligible en fonction de son type de cancer spécifique. « Les résultats ont été assez frappants », a déclaré Caulfield. Par exemple, ils ont découvert que 94 pour cent des tumeurs de patients atteints de glioblastome multiforme, une forme agressive de cancer du cerveau, présentaient de petites variantes dans au moins un gène, et 58 pour cent contenaient un CNA dans au moins un gène. Les panels ciblés standard de soins ne testent pas la plupart des mutations observées. « Cette architecture génétique complexe a conduit à la conclusion que pour caractériser réellement la variation génétique du gliome, un génome entier serait le meilleur test », a déclaré Caulfield.
Le gène le plus fréquemment muté parmi les types de cancer était la protéine tumorale 53 (TP53). Cependant, au sein de chaque type de cancer, la survenue de chaque mutation était variable, mise en évidence par une fréquence élevée de TP53 SNV dans le carcinome séreux ovarien de haut grade (90 pour cent) et une faible fréquence de la mutation dans le mésothéliome (sept pour cent).
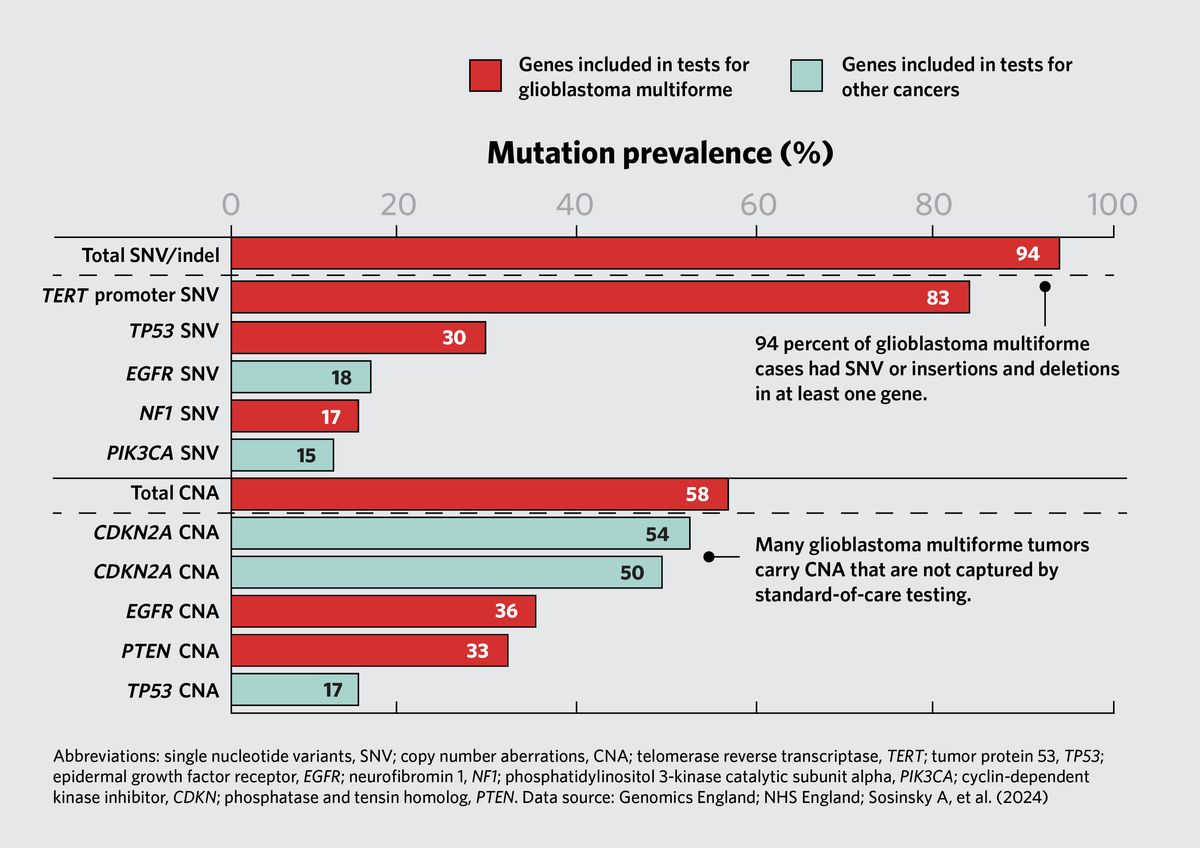
Le séquençage du génome entier capture la complexité génétique du glioblastome multiforme. Les données sont basées sur les gènes indiqués pour les tests dans le National Genomic Test Directory for Cancer. L’image comprend les cinq principaux taux de mutation du SNV et du CNA pour le type de cancer.
Érin Lemieux
L’équipe de recherche a également évalué les taux de mutation des gènes qui ne sont pas inclus dans le protocole de test standard d’un type de cancer. Par exemple, 54 pour cent des carcinomes de l’endomètre du corps utérin et 49 pour cent des adénocarcinomes endométrioïdes de l’ovaire portaient une sous-unité catalytique alpha de la phosphatidylinositol 3-kinase (PIK3CA) mutation. Cependant, dans le système de santé britannique, cette mutation n’est testée que dans les carcinomes invasifs du sein. Ces résultats suggèrent que les scientifiques pourraient tester le potentiel thérapeutique de PIK3CA inhibiteurs dans les essais cliniques pour ces cancers.
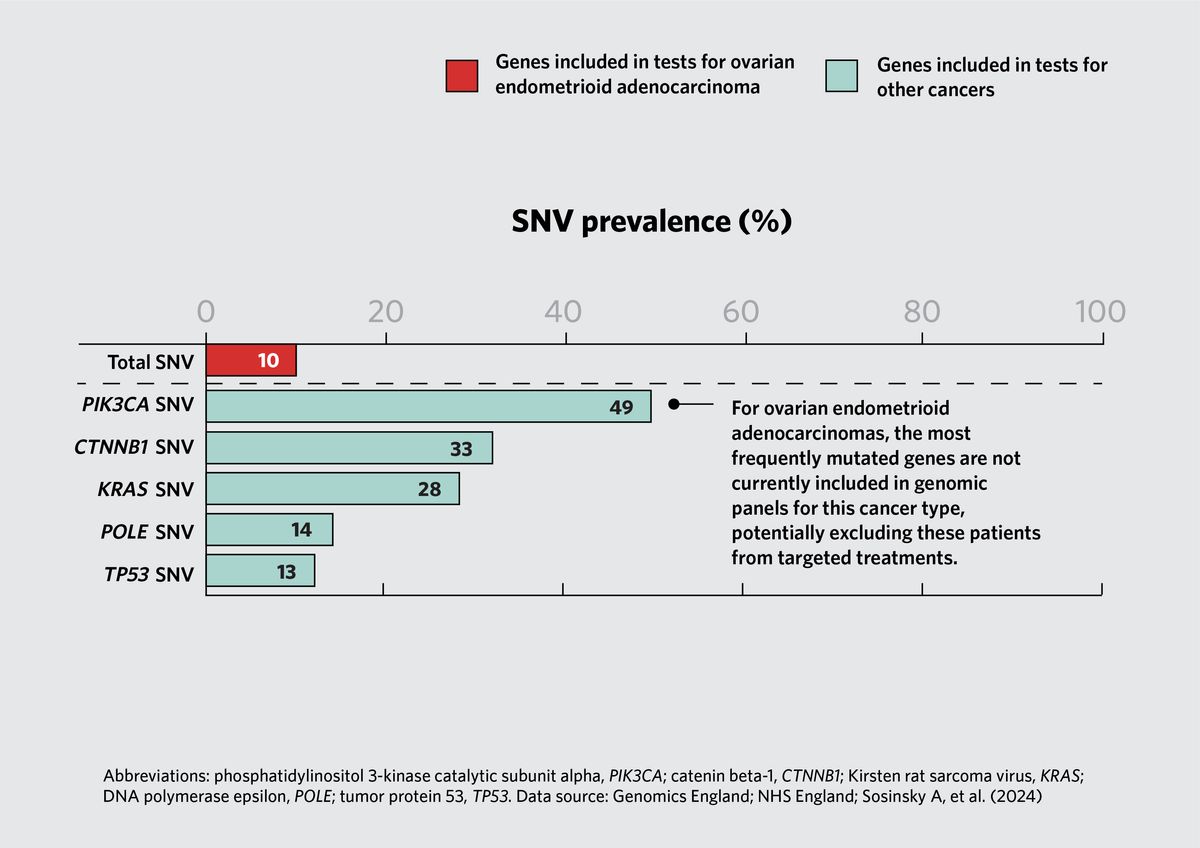
Le séquençage du génome entier révèle des mutations exploitables dans les adénocarcinomes endométrioïdes ovariens. Les données sont basées sur les gènes indiqués pour les tests dans le National Genomic Test Directory for Cancer. L’image comprend les cinq principales variantes nucléotidiques (SNV) pour le type de cancer.
Érin Lemieux
Le déficit de recombinaison homologue (HRD) se produit lorsque les cellules ne parviennent pas à réparer les cassures double brin de l’ADN via la recombinaison homologue. Caulfield et son équipe ont découvert que 40 pour cent des carcinomes séreux ovariens de haut grade avaient des gènes avec HRD. Lorsqu’ils ont incorporé les données cliniques des patients dans leur analyse, ils ont constaté que les patients dont les tumeurs présentaient des taux élevés de HRD obtenaient de meilleurs résultats avec les chimiothérapies à base de platine par rapport aux patients ne présentant aucune altération génétique des gènes de la voie HR ; cela était particulièrement vrai pour les patientes atteintes de carcinomes ovariens de haut grade.
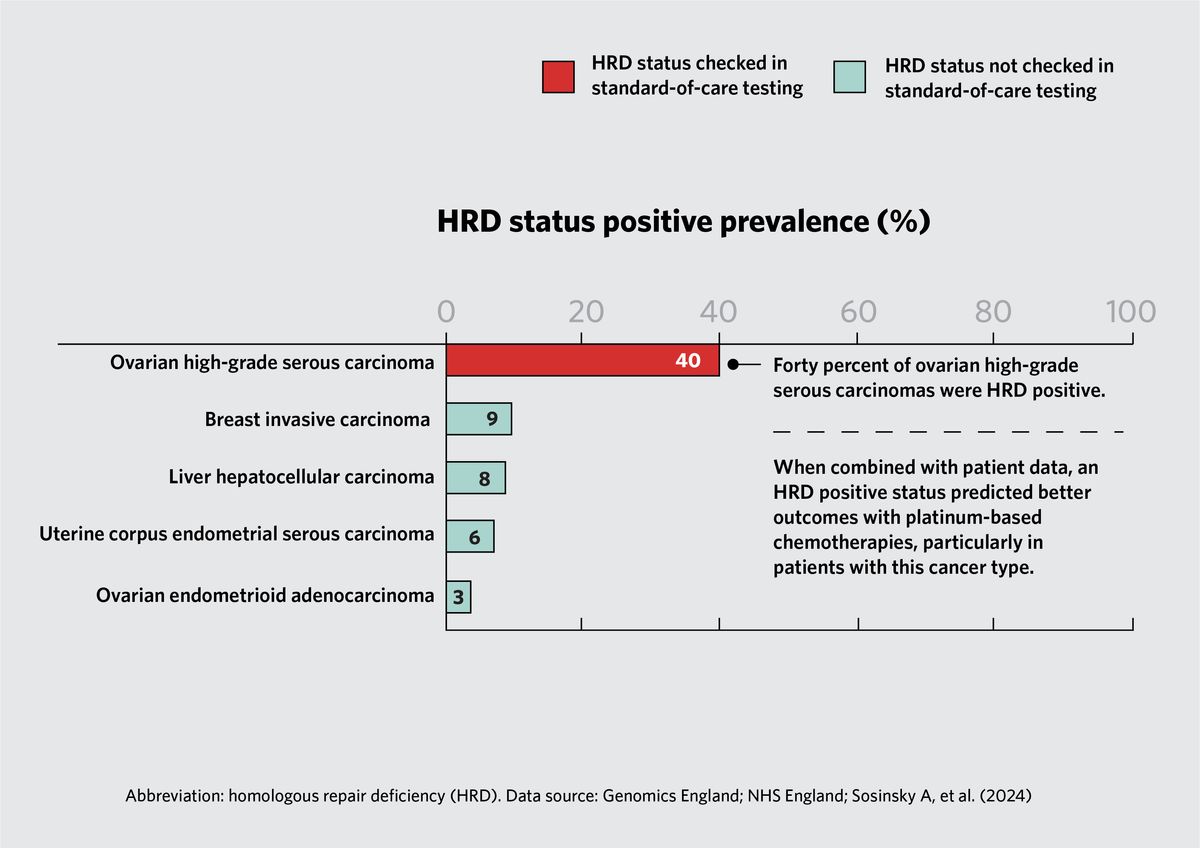
Les marqueurs pangénomiques fournissent des biomarqueurs prédictifs. Les données sont basées sur les gènes indiqués pour les tests dans le National Genomic Test Directory for Cancer.
Érin Lemieux
Parce que l’équipe de recherche a utilisé une approche WGS intégrée, elle a pu séquencer l’ADN tumoral et non tumoral du patient en tandem à la recherche de variantes contenant des informations sur la prédisposition du patient à la malignité pouvant guider sa gestion clinique. Par exemple, 13 pour cent des patientes atteintes d’un carcinome séreux de l’ovaire de haut grade présentaient des variantes BRCA1 et BRCA2 gènes. Les patients atteints de ce type de cancer présentaient un cancer plus précoce s’ils étaient porteurs de variantes germinales prédisposantes. « Si nous pouvions exploiter ces informations dans les familles présentant de forts antécédents de cancer, nous serions peut-être en mesure de faire un meilleur travail de dépistage et de concentrer le dépistage sur des individus spécifiques », a déclaré Caulfield.
L’étude actuelle incluait uniquement les données WGS et le pipeline ne tenait donc compte que du profil génomique d’un patient. « Il aurait été intéressant d’inclure le séquençage de l’ARN ou le séquençage du transcriptome, car cela vous donne un cadre épigénétique pour le microenvironnement tumoral », a déclaré Chinnaiyan. « Il leur manque en quelque sorte l’aspect fonctionnel du génome qu’apporte le séquençage de l’ARN. »
Quoi qu’il en soit, il a noté que les découvertes génomiques étaient impressionnantes et intrigantes. « (L’étude) va avoir un impact sur la communauté de la recherche sur le cancer », a déclaré Chinnaiyan, dont le groupe de recherche reçoit déjà des accès aux données pour soutenir les études en cours.
Lorsque le projet 100 000 génomes a démarré en 2013, les tests génétiques pour le cancer étaient limités. WGS a révolutionné la recherche sur le cancer, en dévoilant l’architecture génétique complexe des cellules cancéreuses ; cependant, cette technologie est largement réservée à la recherche.
« L’avenir des soins contre le cancer impliquera presque certainement un certain degré de profilage génomique, voire multiomique, qui nous permettra de choisir plus correctement les thérapies pour les patients », a déclaré Caulfield. Chinnaiyan considère également que WGS joue un rôle majeur dans les soins contre le cancer du futur. « La vision à long terme de l’oncologie de précision est de mettre en œuvre cette approche globale si les coûts peuvent être gérés », a déclaré Chinnaiyan. Bien que le coût du séquençage d’un génome humain entier ait considérablement diminué depuis l’époque du Projet Génome Humain, il reste toujours prix prohibitif une fois les coûts de traitement, d’analyse, d’interprétation et de stockage être pris en compte.4,5
Cependant, Caulfield espère que ce jour viendra bientôt. Il a déclaré : « À terme, ce qui va arriver, c’est que le prix d’un génome entier va baisser, et à ce stade, il est très probable que tout le monde s’orientera vers le séquençage du génome entier parce que ce sera rentable. Et cela nous permettra de détecter des caractéristiques inhabituelles dans les cancers humains qui vont au-delà de nos connaissances actuelles.
Caulfield a déclaré que la prochaine phase consiste à associer le WGS aux essais cliniques pour comprendre s’ils peuvent identifier des biomarqueurs pour la réponse au traitement. Avec l’architecture mutationnelle d’une tumeur et des données cliniques électroniques en main, les chercheurs pourraient suivre le parcours de santé d’un patient au-delà de la fin d’un essai clinique.
Les références
- Sosinsky A, et al. Aperçus pour l’oncologie de précision grâce à l’intégration des données génomiques et cliniques de 13 880 tumeurs du programme 100 000 Genomes Cancer. Nat Med. 2024;30(1):279-289.
- Turnbull C, et al. Le projet 100 000 génomes : amener le séquençage du génome entier au NHS. BMJ. 2018;361:k1687.
- Kurian AW, et al. Tests génétiques germinaux après diagnostic de cancer. JAMA. 2023;330(1):43-51.
- Akhoundova D, Rubin MA. Le grand défi de déplacer le séquençage du génome entier du cancer vers la clinique. Nat Med. 2024;30(1):39-40.
- Schwarze K, et al. Les coûts complets du séquençage du génome : une étude microcostique sur le cancer et les maladies rares provenant d’un seul centre au Royaume-Uni. Genet Med. 2020;22(1):85-94.



