UNL’ovaire ne mesure que quelques centimètres de large, mais son précieux contenu est encore plus petit. Les ovocytes mesurent quelques dizaines de micromètres de diamètre et sont enveloppés individuellement dans des couches de cellules spécialisées qui forment un follicule qui soutient l’ovule en développement. « (L’ovaire) dicte l’avenir de notre espèce », a déclaré Jun Libioinformaticien à l’Université du Michigan.
Un ovaire peut contenir seulement dix follicules à la fois, ce qui constitue un obstacle à l’étude de ces cellules. Même avec des technologies à haute résolution comme le séquençage d’ARN unicellulaire (scRNA-seq), cellules folliculaires clés peut être difficile à isoler.1,2
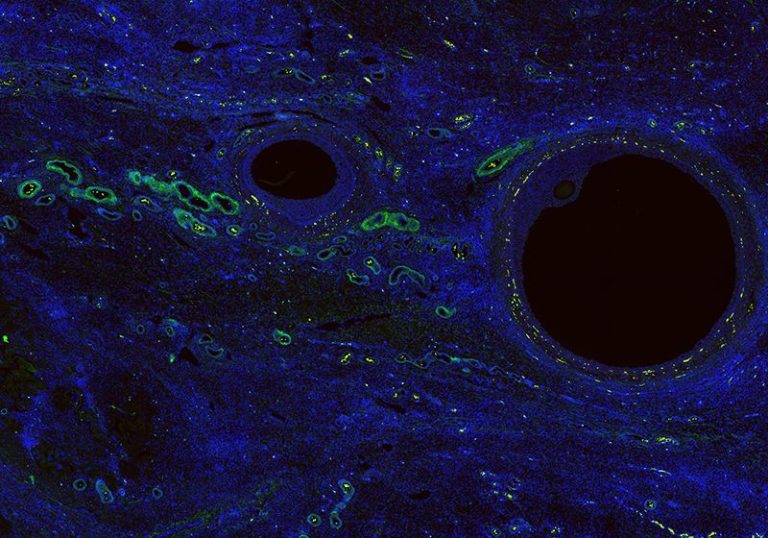
Pour examiner de plus près ces cellules insaisissables, Li s’est associé à Ariella Chikanovbio-ingénieur à l’Université du Michigan, pour étudier ovaires sains en combinant les technologies de profilage spatial et unicellulaire.3 Dans un article publié dans Avancées scientifiques, ils ont présenté un atlas détaillé de l’ovaire, et comme des plans de ville, ils ont zoomé sur les follicules pour observer de plus près les cellules rares. La capture des modèles d’expression des gènes folliculaires peut aider les scientifiques à identifier les facteurs qui favorisent la santé des ovules.
« La morphologie de l’ovaire est si belle », a déclaré Shikanov. « En combinant (spatial et scRNA-seq), nous avons pu construire un atlas plus nuancé. »
L’équipe a étudié deux ovaires de femmes jeunes et en bonne santé – une rareté pour les biologistes de la reproduction, puisque la plupart des ovaires disponibles pour la recherche proviennent de personnes atteintes de maladies de la reproduction ou ménopausées, a déclaré Produits Brittun bio-ingénieur du Dartmouth College qui n’a pas participé à cette étude.
À partir de tranches d’ovaires, les chercheurs ont sélectionné plus de 200 caractéristiques intéressantes, notamment les follicules anciens et jeunes, les couches spécifiques de cellules à l’intérieur des follicules et la surface de l’ovaire. Ensuite, en utilisant une plateforme de biologie spatiale appelée GeoMx, ils ont mesuré les niveaux d’ARN de plus de 18 000 gènes. Bien que cette technologie n’ait pas de résolution monocellulaire, elle a balayé la région sélectionnée et capturé l’expression de l’ARN de petits groupes d’environ une douzaine de cellules adjacentes. Les chercheurs ont utilisé ces informations pour cartographier les modèles spatiaux d’expression des gènes dans les cellules qui n’avaient pas été bien décrits auparavant.
À l’intérieur des follicules, ils ont trouvé non seulement des ovocytes, mais également des cellules de la thèque et de la granulosa, deux types de cellules sécrétant des hormones qui entourent les œufs immatures. En fouillant dans les profils d’expression génique de ces cellules, les chercheurs ont compilé des listes de gènes que d’autres chercheurs peuvent désormais utiliser pour identifier ces cellules dans leurs propres ensembles de données. L’étude a également offert des informations sur la biologie ovarienne. Par exemple, lorsque les auteurs ont mesuré les gradients d’expression des gènes qui se produisaient dans les anneaux concentriques autour des follicules, ils ont découvert que les gènes codant pour les signaux favorisant le développement des ovocytes étaient fortement exprimés dans les noyaux des follicules, tandis que les gènes codant pour les signaux favorisant la production d’hormones et l’ovulation étaient concentrés dans la périphérie.
Avec trois ovaires différents, les chercheurs ont utilisé scRNA-seq pour mesurer l’expression des gènes de manière impartiale. « Le mariage des deux technologies peut s’avérer très utile, car vous pouvez voir exactement où un groupe particulier de cellules est important dans les tissus », a déclaré Goods, même si elle a noté qu’avec un échantillon de petite taille et une technologie spatiale relativement grossière, elle aimerait voir validation des types cellulaires et de leurs profils d’expression génique dans des études futures. « Cet article jette vraiment les bases de nombreuses études futures dans ce domaine, ce qui est vraiment passionnant », a-t-elle déclaré.
Li et Shikanov pensent que d’autres chercheurs peuvent utiliser cet atlas comme référence pour identifier les types de cellules et les régions clés dans d’autres études ovariennes, ou comme point de comparaison pour trouver des voies moléculaires dérégulées dans la maladie. Shikanov voit également le potentiel de cet atlas pour promouvoir le développement de traitements contre la fertilité. Elle souhaite développer des ovaires artificiels et peut désormais utiliser l’atlas pour déterminer quels gènes sont essentiels aux cellules qui soutiennent le développement des ovules.
L’atlas des cellules ovariennes ne marque pas la fin de la collaboration entre Shikanov et Li. Ce projet fait partie d’un projet en trois parties qu’ils entreprennent au nom du Human Cell Atlas, un consortium international qui vise à cartographier tous les organes du corps humain à une résolution unicellulaire. Le duo a précédemment publié une étude sur trompes de Fallopeet ont désormais jeté leur dévolu sur le utérus.4,5 Ils espèrent également isoler des ovocytes individuels et les suivre tout au long de leur développement afin de trouver les gènes qui orchestrent l’ovulation.
« Il existe différents types d’infertilité, donc connaître la biologie nous donne une meilleure base pour prédire et intervenir », a déclaré Li.
Les références
1. Fan X, et coll. Reconstruction unicellulaire du remodelage folliculaire dans l’ovaire adulte humain. Nat Commun. 2019;10(1):3164.
2. Wagner M, et coll. L’analyse unicellulaire du cortex ovarien humain identifie des populations cellulaires distinctes mais aucune cellule souche oogoniale. Nat Commun. 2020;11(1):1147.
3. Jones ASK, et al. Atlas cellulaire de l’ovaire humain utilisant la transcriptomique spatiale guidée morphologiquement et le séquençage unicellulaire. Avancé en sciences. 2024;10(14):EADM7506.
4. Ulrich ND, et al. Hétérogénéité cellulaire des trompes de Fallope humaines dans des états pathologiques normaux et hydrosalpinx identifiées à l’aide de scRNA-seq. Cellule de développement. 2022;57(7):914-929.e7.
5. Ulrich ND, et al. Hétérogénéité cellulaire et dynamique de l’utérus humain chez les femmes préménopausées en bonne santé. bioRxiv. 2024.