CLes ancêtres surviennent généralement lorsque les cellules accumulent des mutations dans leur ADN qui les empêchent de contrôler la division cellulaire.1 Cependant, pour certains types de tumeurs, les chercheurs ont j’ai eu du mal à trouver des mutationsce qui amène les scientifiques à s’interroger sur leurs causes.2 Or, dans une étude publiée dans Nature, Les chercheurs ont découvert que des changements épigénétiques de courte durée peuvent modifier de manière permanente l’expression des gènes et déclencher le cancer.3 Alors que la plupart des cancers se développent à la suite de mutations, leurs résultats suggèrent que quelques types de tumeurs pourraient déroger à cette règle.
Pendant des années, Giacomo Cavalligénéticien au Centre national de la recherche scientifique, et ses collègues ont étudié le rôle que jouent les facteurs épigénétiques appelés Protéines polycomb jouer dans le cancer.4 Ces protéines forment des complexes qui enroulent la chromatine et désactivent les gènes qui favorisent la division cellulaire. L’équipe a précédemment découvert que mutations dans les facteurs Polycomb provoquent un démêlage de la chromatine, qui se traduit par une prolifération cellulaire et l’apparition du cancer.5 Ils se sont demandé s’ils pourraient obtenir le même effet en désactivant temporairement les gènes Polycomb.
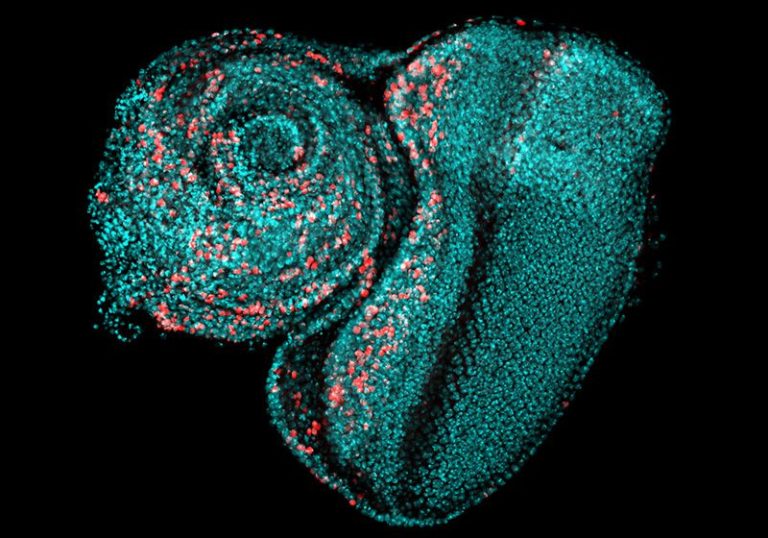
Pour tester leur hypothèse, ils se sont tournés vers la mouche à fruits, Drosophila melanogaster; l’espèce ne possède qu’une seule copie de chaque gène impliqué dans la machinerie Polycomb, ce qui facilite la perturbation du système. Les protéines Polycomb jouent un rôle clé au cours du développement en influençant le moment de la différenciation cellulaire. Cavalli et son équipe ont étudié l’impact de la perte de ce contrôle épigénétique sur les structures larvaires précoces appelées disques imaginaux. À l’aide d’un système d’interférence ARN sensible à la température, ils ont exposé les disques à des températures plus élevées pendant 24 heures, ce qui a temporairement désactivé les gènes Polycomb pendant deux jours.
« Ils ont très bien montré qu’avec ce système transitoire, ils pouvaient désactiver brièvement ce gène de développement, le réactiver, et cela suffisait à déclencher la tumorigenèse », a déclaré Douglas Hanahanun biologiste spécialisé dans le cancer à l’École polytechnique fédérale de Lausanne, qui n’a pas participé aux travaux.
Pour s’assurer que les mutations génétiques ne déclenchent pas ces cancers, l’équipe a séquencé les cellules cancéreuses en même temps que des témoins sains. « Il y a effectivement des mutations », a déclaré Cavalli, « mais il n’y a aucune différence dans la quantité d’événements mutationnels dans les échantillons cancéreux par rapport aux échantillons témoins. »
Pour confirmer que ces mutations n’étaient pas à l’origine des cancers, l’équipe a suivi la position des cellules tumorales dans les disques imaginaux. Ils ont émis l’hypothèse que si les mutations donnaient naissance à un cancer, elles auraient commencé avec une seule cellule fautive, tandis que la reprogrammation épigénétique aurait provoqué la rébellion de l’ensemble du tissu. Pour repérer les cellules tumorales à division rapide, ils ont coloré les disques imaginaux avec de la 5-éthynyl-2′-désoxyuridine (EdU), un colorant qui prend la place de la thymine lors de la réplication de l’ADN.6 L’EdU était omniprésent dans tous les disques imaginaux, indiquant que la reprogrammation épigénétique à l’échelle des tissus était le coupable du cancer.
L’équipe de Cavalli a découvert que la suppression temporaire des facteurs Polycomb activait les gènes qu’ils répriment généralement. Cela a conduit à une augmentation de la signalisation des transducteurs de signal et activateurs de transcription Janus kinase (JAK-STAT), ce qui favorise la prolifération cellulaire.7 L’activation de JAK-STAT a également provoqué un effet domino, en faisant basculer l’interrupteur sur d’autres gènes liés au cancer. Cela comprenait le démarrage du Homéodomaine-1 à doigt de zinc gène, qui empêche les cellules en développement de se différencier et ralentit finalement la croissance.8
Selon Cavalli, l’étude soulève des questions sur la fréquence à laquelle les altérations épigénétiques entraînent la formation de tumeurs. « Cela peut être plus courant qu’on ne le pense », a déclaré Cavalli. « Lorsque nous séquençons des cancers et découvrons des mutations, nous le faisons généralement lorsque le cancer est déjà développé. » Cependant, il est possible que des forces épigénétiques aient transformé les cellules initiales, a-t-il suggéré. En effet, cela pourrait s’appliquer à certains cancers du cerveau chez les enfants.9 « Il n’existe aucune preuve d’une avalanche de mutations dans ces tumeurs pédiatriques, et pourtant il existe des tumeurs », a déclaré Hanahan, ajoutant que ces types de cancer pourraient être un terrain fertile pour explorer les déclencheurs épigénétiques chez l’homme.
Les chercheurs ne savent pas exactement quels facteurs déclenchent ces changements épigénétiques de courte durée, à l’origine du cancer. « L’inflammation est un facteur majeur de l’initiation des tumeurs », a déclaré Hanahan. Les cellules immunitaires pourraient sécréter un milieu de signaux chimiques qui perturbent le contrôle épigénétique de l’expression des gènes dans les cellules et déclenchent des cancers, a-t-il suggéré.
À l’avenir, Cavalli souhaite s’éloigner de la mouche à fruits pour étudier les facteurs épigénétiques dans des modèles de mammifères qui ont une réponse immunitaire et une vascularisation – qui influencent toutes deux l’évolution du cancer – qui reflètent mieux celles des humains.
Cavalli a noté que certains chercheurs développent thérapies épigénétiques contre le cancerqui visent à inverser les cellules cancéreuses au lieu de les tuer.dix Des recherches plus poussées sur le rôle de l’épigénétique dans l’initiation des tumeurs pourraient aider les développeurs de médicaments à déterminer comment maîtriser ces facteurs de cancer longtemps négligés.
Les références
1. Hanahan D. Les caractéristiques du cancer : de nouvelles dimensions. Découverte du cancer. 2022;12(1):31-46.
2. McDonald OG, et al. La reprogrammation épigénomique au cours de la progression du cancer du pancréas relie le métabolisme anabolique du glucose aux métastases à distance. Nat Genet. 2017;49(3):367-376.
3. Parreno V, et al. La perte transitoire des composants de Polycomb induit un destin épigénétique cancéreux. Nature. 2024;629(8012):688-696.
4. Parreno V, et al. Mécanismes de la fonction des protéines du groupe Polycomb dans le cancer. Cellule Res. 2022;32(3):231-253.
5. Loubière V, et al. Le redéploiement coordonné des protéines PRC1 supprime la formation de tumeurs pendant Drosophile développement. Nat Genet. 2016;48(11):1436-1442.
6. Flomerfelt FA, Gress RE. Analyse de la prolifération et de l’homéostasie cellulaires à l’aide du marquage EdU. Dans : Bosselut R, S. Vacchio M, éd. Développement des cellules T. Méthode Mol Biol. 2016;1323:211-220.
7. Zoranovic T, et al. Régulation de la prolifération, de la compétition cellulaire et de la croissance cellulaire par le Drosophile Voie JAK-STAT. JAK-STAT. 2013;2(3):e25408.
8. Leatherman JL, DiNardo S. Zfh-1 contrôle l’auto-renouvellement des cellules souches somatiques dans le Drosophile testicule et influence de manière non autonome l’auto-renouvellement des cellules souches germinales. Cellule souche. 2008;3(1):44-54.
9. Jenseit A, et al. EZHIP : une nouvelle pièce du puzzle pour comprendre l’épendymome de la fosse postérieure chez l’enfant. Acta Neuropathol. 2022;143(1):1-13.
10. Miranda Furtado CL, et al. Epidrugs : cibler les marques épigénétiques dans le traitement du cancer. Épigénétique. 2019;14(12):1164-1176.